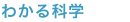分析法の改良によって環境DNAを系統地理学の研究に利用できることを確認
(2023年4月01日)
同じ種類の野生生物でも生息地域によってDNAに記された遺伝情報は微妙に違っていて、この違いを利用して生物種の分布や進化の過程などを明らかにしようとする研究は「系統地理学」と呼ばれています。しかし、地域ごとに遺伝情報を比較するには、数多くの調査地点で生物を捕獲し、解読するDNAを得るための組織サンプルを採取しなければならず、多大な労力と時間を要していました。そこで野生生物の遺伝子を調べるのに、近年、注目を集めているのが環境中に存在する「環境DNA」です。
野生生物が生息する環境には、体から剥がれ落ちた皮膚や排泄された糞などに含まれているDNAが存在するはずで環境DNAと呼ばれています。水棲生物の場合、環境DNAは水中を漂うことになり、調査地点の池や川で採取した水を分析するだけで研究対象の生物の有無を確かめられるので、様々な生物の調査に環境DNAが応用されるようになっています。
ただし、特定の生物がいるかどうかを確かめるのと異なり、同じ種類の中で生じるわずかな差異を環境DNAで捉えることは難しいと考えられてきました。そこで京都大学、福岡工業大学、山口大学などが参加した研究グループは、過去の研究で実際に捕獲して、地域ごとのDNAの違いが明らかになっている淡水魚5種(ドンコ、イシドンコ、カワムツ、ヌマムツ、オイカワ)を対象にして環境DNAの分析法の改良に取り組みました。
研究対象とした5種が自然に分布している西日本の広範囲の河川で、過去に採集して冷凍保存していたサンプルを含めて、合計94地点で川の水を採取(図1)。そこから環境DNAを抽出しました。新型コロナウイルス感染症の診断にも用いられているPCR法により、細胞内小器官の三コトンドリアに含まれるDNAを増幅して、その塩基配列を解読しました。ただし、得られた遺伝情報の中にはノイズが含まれているため、非常に稀な塩基配列を排除などのデータスクリーニングを3段階で行いました。
排除されることなく残った塩基配列を、様々な生物の遺伝情報を集めた国際DNAデータベースに登録されている配列と完全一致するかどうかを調べたところ、段階が進むごとに一致する割合が上昇したことで、試されたデータスクリーニングが妥当なものであることが確認されました。
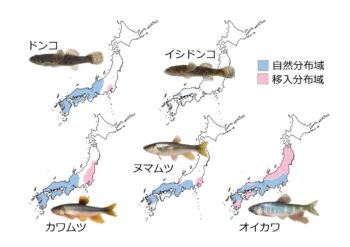
3段階でスクリーニングされてもなお、排除されることなく残った塩基配列を使って、塩基配列の違いを樹木の枝分かれのような形で示した分子系統樹を推定して、従来通りの捕獲調査によって作成された分子系統樹と比較したところ、ほぼ完全に一致することが分かりました。ドンコ類に関しては、捕獲調査では九州東部に未知の系統が存在することが示唆されたため、改めて捕獲調査を行い、新たな系統の存在が確認されました(図2)。
今回の研究では地域ごとに遺伝子の違いが明らかになっている淡水魚5種を対象に行われましたが、分析法次第で環境DNAを使えることが明らかになったことで、今後、環境DNAの活用で系統地理学の研究が進むと期待されます。
【参考】
■京都大学プレスリリース

斉藤 勝司(さいとう かつじ)
サイエンスライター。大阪府出身。東京水産大学(現東京海洋大学)卒業。最先端科学技術、次世代医療、環境問題などを取材し、科学雑誌を中心に紹介している。